В исследованиях рака модели искусственного искусства учатся видеть, что могут пропустить ученые
18 июля 2025 г.Таблица ссылок
Аннотация и I. Введение
Материалы и методы
2.1. Несколько экземпляров обучения
2.2. Модель архитектуры
Результаты
3.1. Методы обучения
3.2. Наборы данных
3.3. WSI предварительно обработайте трубопровод
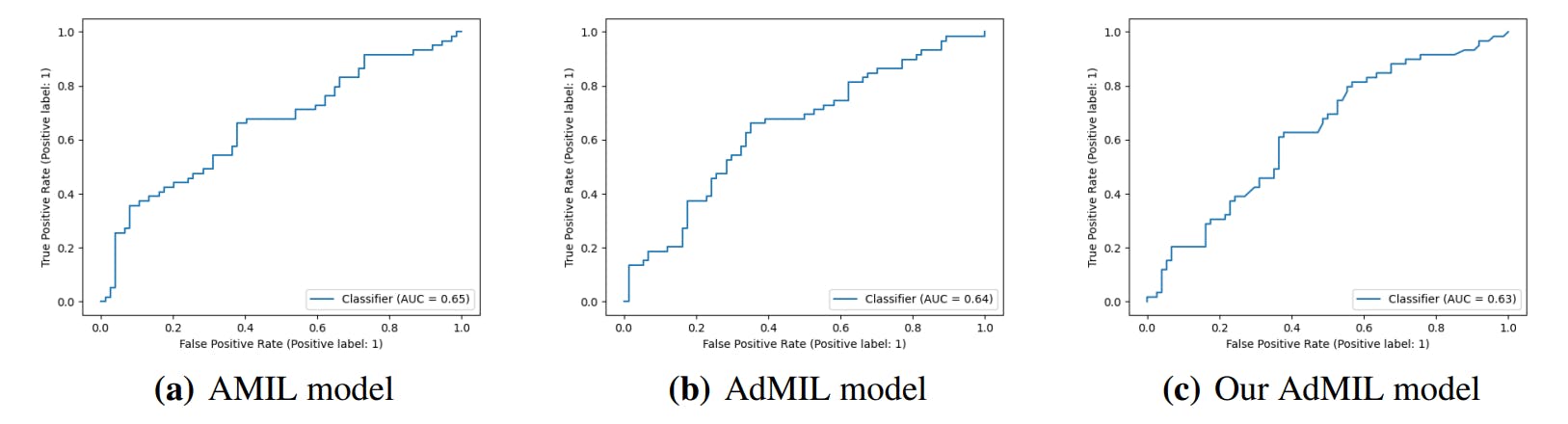
3.4. Результаты классификации и обнаружения ROI
Дискуссия
4.1. Задача обнаружения опухоли
4.2. Задача обнаружения мутаций генов
Выводы
Благодарности
Авторская декларация и ссылки
5. Выводы
В этой работе мы изучаем характеристики рамок обучения с несколькими инстанциями (MIL) с механизмами внимания для классификации WSI и виртуальным окрашиванием опухолей молочной железы. Мы исследовали два различных подхода, которые идентифицируют различные морфологические прокси для классификаторов патча (плитка). Эти две рамки были использованы в слаборевенемом применении к обнаружению опухоли и обнаружению мутаций TP53 при легком раке молочной железы и плоскоклеточном раке. Мы обнаружили, что было гораздо легче идентифицировать интересующие области (ROI), распознавая опухоль против непуходов даже при низком разрешении (AUC> 0,95), чем для классификации TP53 MTITED по сравнению с не-мутированным (AUC <0,71). Наблюдение о том, что более высокие резолюции (20x) работали лучше, чтобы идентифицировать рентабельность инвестиций для мутации, само по себе не было неожиданностью, но возможности для гипотезы о новых морфологических интерпретациях стали основным результатом работы здесь. Точно так же, когда мы исследовали новые модификации установленного метода обучения с несколькими инстанциями, изменяя его первоначальный слой внимания, наиболее интересным результатом была не улучшенная точность, а внимание, которое модель уделяет различным морфологическим особенностям (то есть «скользящая живопись»). В частности, результаты, описанные на рисунках 3, 4 и 5, иллюстрируют возможности для интерактивного исследования повторяющихся морфологий для их роли в этиологии рака.
6. Благодарности
Эта работа была частично профинансирована Национальным институтом рака (NCI) Программа исследований CAS#10901 (Episphere), Фондом восстановления и устойчивости к Центру ответственного ИИ (ссылка C628696807-00454142) и финансирование Фонда по науке и технологии (FCT) для Inescid (REF. Uidb/5001/2020).
Открытый исходный код, используемый в этом проекте, общедоступен по адресу https://github.com/timafonso/wsi_mil_roi
7. Авторская декларация
Нет конфликта интересов, чтобы сообщить.
Ссылки
[1] Bahdanau, D., Cho, K.H., Bengio, Y., 2015. Перевод нейронной машины путем совместного обучения для выравнивания и перевода. 3 -я Международная конференция по обучению, ICLR 2015 - Требование конференции, 1–15.
[2] Dietterich, T.G., Lathrop, R.H., Lozano-Pérez, T., 1997. Решение задачи множественного экземпляра с оси-параллельными прямоугольниками. Искусственный интеллект 89, 31–71.
[3] Dimitriou, N., Arandjelović, O., Caie, P.D., 2019. Глубокое обучение для анализа изображений всего слайда: обзор. Границы в медицине 6, 1–7.
[4] Guo, B., Li, X., Yang, M., Zhang, H., Xu, X.S., 2023. Надежный и легкий алгоритм обучения с множественным вниманием для прогнозирования генетических изменений. Компьютерная медицинская визуализация и графика 105, 102189.
[5] Hanna, M.G., Parwani, A., Sirintrapun, S.J., 2020. Визуализация целого слайда: технология и приложения. Достижения в анатомической патологии 27, 251–259.
[6] Hou, W., Yu, L., Lin, C., Huang, H., Yu, R., Qin, J., Wang, L., 2022. H2-Mil: Изучение иерархического представления с гетерогенным изучением множественного экземпляра для всего слайда. Труды конференции АААИ по искусственному интеллекту 36, 933–941.
[7] Ilse, M., Tomczak, J., Welling, M., 2018. Основанное на внимании обучение глубокому множеству инстанций, в: Международная конференция по машинному обучению, с. 2127–2136.
[8] Javed, S.A., Juyal, D., Padigela, H., Taylor-Weiner, A., Yu, L., Prakash, A., 2022. Аддитивное MIL: по сути интерпретируемое обучение множественным экземплярам для патологии. Достижения в системах обработки нейронной информации 35, 20689–20702.
[9] Хенден, М., Кори, А., Раджкумар, Х., Кришнамурти, Г., Сринивасан, Б., 2021. Обобщенная структура глубокого обучения для сегментации и анализа изображений. Scientific Reports 11, 1–14.
[10] Konstantinov, A.V., Utkin, L.V., 2022. Многоподобное обучение множественным экземплярам. Нейронные вычисления и приложения, 1–23.
[11] Li, B., Li, Y., Eliceiri, K.W., 2021. Несета обучения с несколькими потоками для цельной классификации изображений слайдов с самоподобным контрастным обучением. Материалы конференции компьютерного общества IEEE по компьютерному зрению и распознаванию шаблонов, 14313–14323.
[12] Li, Z., Yuan, L., Xu, H., Cheng, R., Wen, X., 2020. Глубокое обучение в области многочисленного инстанции с индуцированным самоуничтожением для классификации медицинских изображений. Труды - 2020 IEEE Международная конференция по биоинформатике и биомедицине, BIBM 2020, 446–450.
[13] Lu, M., Pan, Y., Nie, D., Liu, F., Shi, F., Xia, Y., Shen, D., 2021a. Улыбка: на основе редкого взаимодействия с множественным контрастным обучением для классификации подтипа глиомы с использованием патологических изображений, в кн.: Семинар MICCAI по вычислительной патологии, стр. 159–169.
[14] Lu, M.Y., Williamson, D.F., Chen, T.Y., Chen, R.J., Barbieri, M., Mahmood, F., 2021b. Данные эффективные и слабо контролируемая вычислительная патология на изображениях целых слайдов. Nature Biomedical Engineering 5, 555–570.
[15] Наяк, Н., Чанг, Х., Боровский, А., Спеллман, П., Парвин, Б., 2013. Классификация гистопатологии опухоли посредством разреженного обучения функции, в: 2013 IEEE 10th Международный симпозиум по биомедицинской визуализации, с. 410–413.
[16] Otsu, N., 1979. Метод выбора порога из гистограмм уровня серого. IEEE транзакции по системам, человеку и кибернетике 9, 62–66.
[17] Reisenbüchler, D., Wagner, S.J., Boxberg, M., Peng, T., 2022. С. 377–386.
[18] Riasatian, A., Babaie, M., Maleki, D., Kalra, S., Valipour, M., Hemati, S., Zaveri, M., Safarpoor, A., Shafiei, S., Afshari, M., Rasoolijaberi, M., Sikaroudi, M., Adnan, M., Shah, S., S., S. Diamandis, P., Pantanowitz, L., Kashani, H., Ghodsi, A., Tizhoosh, H., 2021. тонкая настройка и обучение Densenet для представления изображения гистопатологии с использованием диагностических слайдов TCGA. Анализ медицинского изображения 70, 102032.
[19] Rymarczyk, D., Borowa, A., Tabor, J., Zielinski, B., 2021. Самоубийство ядра для слабоудовлетворенной классификации изображений с использованием глубокого обучения множеству экземпляров. Труды - 2021 IEEE Winter Conference по приложениям Computer Vision, WACV 2021, 1720–1729.
[20] Shao, Z., Bian, H., Chen, Y., Wang, Y., Zhang, J., Ji, X., et al., 2021. Transmil: Трансформатор -коррелированное изучение множественных экземпляров для классификации изображений цельной слайды. Достижения в системах обработки нейронной информации 34, 2136–2147.
[21] Шейх, Т.С., Ким, Дж.Ю., Шим, Дж., Чо, М., 2022. Неконтролируемое обучение на основе нескольких дескрипторов для диагноза WSIS. Диагностика 12, 1480.
[22] Tomczak, K., Czerwińska, P., Wiznerowicz, M., 2015. Атлас генома рака (TCGA): неизмеримый источник знаний. Wspolczesna onkologia 1a, A68 - A77.
[23] Wang, X., Chen, H., Gan, C., Lin, H., Dou, Q., Tsougenis, E., Huang, Q., Cai, M., Heng, P.A., 2020. Слабо контролировал глубокое обучение для цельного анализа изображения рака легких. IEEE транзакции на кибернетике 50, 3950–3962.
[24] Wang, X., Zou, C., Zhang, Y., Li, X., Wang, C., Ke, F., Chen, J., Wang, W., Wang, D., Xu, X., et al., 2021. Предсказание мутации гена BRCA при раке молочной железы на основе глубокого обучения и изображений гистопатологии. Границы в генетике 12, 661109.
[25] Yao, J., Zhu, X., Jonnagaddala, J., Hawkins, N., Huang, J., 2020. Прогнозирование выживания рака на основе изображений на основе слайдов с использованием внимания, направляемого с сетью обучения в глубоком множестве экземпляров. Анализ медицинского изображения 65.
[26] Zhao, L., Yuan, L., Hao, K., Wen, X., 2022. Общее обучение на основе глубокого многооткрытия. Мультимедийные системы, 1–13.
[27] Zhu, X., Yao, J., Zhu, F., Huang, J., 2017. Труды - 30 -я конференция IEEE по компьютерному зрению и распознаванию образцов, CVPR 2017 2017 -янюа, 6855–6863.
Авторы:
(1) Мармим Афонсо, институт Superior Técnico, Universidade de Lisboa, Av. Ровиско Паис, Лиссабон, 1049-001, Португалия;
(2) Praphulla M.S. Bhawsar, Отдел эпидемиологии и генетики рака, Национальный институт рака, Национальный институт здравоохранения, Bethesda, 20850, штат Мэриленд, США;
(3) Monjoy Saha, Отделение эпидемиологии и генетики рака, Национальный институт рака, Национальный институт здравоохранения, Bethesda, 20850, штат Мэриленд, США;
(4) Джонас С. Алмейда, Отделение эпидемиологии и генетики рака, Национальный институт рака, Национальный институт здравоохранения, Бетесда, 20850, штат Мэриленд, США;
(5) Арлиндо Л. Оливейра, Институт Верхний Течнико, Университет де Лисбоа, ав. Rovisco Pais, Лиссабон, 1049-001, Португалия и INESC-ID, R. Alves Redol 9, Lisbon, 1000-029, Португалия.
Эта статья есть
Оригинал

