Как мы обучали модели ИИ для обнаружения опухолей и генных мутаций
17 июля 2025 г.Таблица ссылок
Аннотация и I. Введение
Материалы и методы
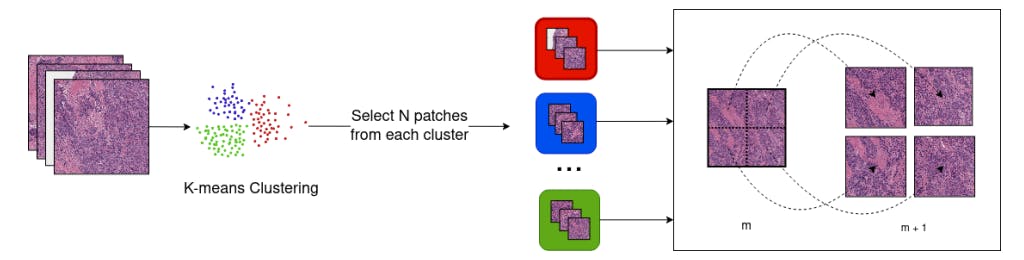
2.1. Несколько экземпляров обучения
2.2. Модель архитектуры
Результаты
3.1. Методы обучения
3.2. Наборы данных
3.3. WSI предварительно обработайте трубопровод
3.4. Результаты классификации и обнаружения ROI
Дискуссия
4.1. Задача обнаружения опухоли
4.2. Задача обнаружения мутаций генов
Выводы
Благодарности
Авторская декларация и ссылки
3. Результаты
3.1. Методы обучения
Мы разделили каждый набор данных на учебный набор (80%) и набор тестов (20%). Для каждой задачи и модели мы выполнили 5-кратную перекрестную проверку на тренировочном наборе, чтобы предотвратить переживание. Затем тестовый набор использовался для внешней проверки, причем модель была извлечена из сгиба, которая получила наилучшие результаты.
Для всех моделей использованной функцией потерь была бинарной потерей перекрестной энтропии, определенной как
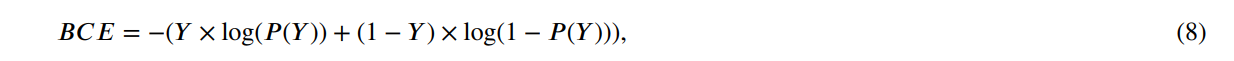
где 𝑌 положительный класс. Для оптимизации потерь мы использовали Adam Optimizer. Для некоторых моделей мы использовали планировщик ставок обучения косинуса.
3.2. Наборы данных
Мы выбрали два проекта из TCGA (атлас генома рака) [22], которые были проанализированы в этой работе: TCGA-BRCA (инвазивная карцинома молочной железы) и TCGA-LUSC (плоскоклеточная карцинома легких).
Для задачи обнаружения опухоли мы использовали только слайды флеш-замороженных. Несмотря на то, что замороженные образцы менее подходят для вычислительного анализа по сравнению с фиксированными формалиновыми слайдами (FFPE), мы решили построить наш набор данных из-за отсутствия слайдов FFPE в TCGA, содержащей только здоровую ткань. Для этой задачи мы сосредоточились на 5 -кратных плитках увеличения, поскольку это уровень увеличения, который обычно используют патологи при поиске опухолей.
Для обнаружения генов мы использовали слайды FFPE, поскольку несбалансированное распределение больше не было проблемой, и эти слайды дали лучшие результаты и результаты обучения. Кроме того, экстрактор особенности, который мы использовали для плиток, Kimianet [18], обучался слайдам FFPE, поэтому соответствие тому же приводит к лучшим результатам. Для этой задачи мы построили три набора данных на трех разных уровнях увеличения: 5x, 10x и 20x, чтобы лучше понять, в каком увеличении модели могут лучше всего обнаружить корреляции между мутацией и морфологией ткани.
Из -за большого размера слайдов FFPE и сэкономить пространство и время хранения, мы выполнили случайную выборку плиток для этих слайдов, в зависимости от уровня увеличения. Более того, в то время как метки присутствия опухоли на уровне слайда были доступны для первой задачи, этикетки экспрессии генов доступны только на уровне случая (уровень пациента), что представляет некоторые проблемы. Мы предположили, что мутация будет присутствовать не только во всех диагностических слайдах от пациента, помеченного как положительный, но и, что она будет охватывать достаточное количество ткани, чтобы быть захваченными в плитках, отобранных в нашем наборе данных.
3.2.1. TCGA-BRCA
TCGA-BRCA состоит из 1098 случаев. Он содержит 1133 слайдов FFPE и слайды Flash-Fresh-Frash-Flash в этих случаях. Этот набор данных использовался для задачи мутации гена. Мы сосредоточились на мутациях гена TP53, так как этот ген показывает большее количество мутированных случаев из тех, кто проверяется на простые соматические мутации (331 из 969 случаев), что позволяет нам создать сбалансированный набор данных. Для этой задачи у нас есть 349 положительных слайдов и 670 отрицательных слайдов. Мы выбрали равное количество положительных и отрицательных WSIS, и после фильтрации неадекватных слайдов по обработке мы получили 662 слайда, 331 помечены положительными и 331 маркированными отрицательными.
3.2.2. TCGA-LUSC
TCGA-LUSC-это набор данных для плоскоклеточного рака легких. Размер этого набора данных довольно мал по сравнению с TCGA-BRCA, с 504 случаями, содержащими 512 диагностических слайдов и 1100 тканевых слайдов в этих случаях. Для задачи обнаружения опухоли мы имеем 753 положительных слайдов и 347 отрицательных слайдов. Его классовое распределение для задачи обнаружения опухоли не слишком несбалансировано для наших целей. Мы выбрали равное количество положительных и отрицательных слайдов, в конечном итоге набором данных, состоящим из 694 слайдов. Его уменьшенное количество слайдов, а также наличие большего количества артефактов делает этот тип рака более сложным для работы. Слайды TCGA-BRCA всегда имеют процент опухоли, по крайней мере, 90%, в то время как в случае TCGA-LUSC распределение процента опухоли более сбалансировано. Поэтому мы решили, что для этой задачи набор данных, построенный со слайдами от TCGA-LUSC, предпочтительнее подтверждения способности модели обобщать с различными слайдами.
Авторы:
(1) Мармим Афонсо, институт Superior Técnico, Universidade de Lisboa, Av. Ровиско Паис, Лиссабон, 1049-001, Португалия;
(2) Praphulla M.S. Bhawsar, Отдел эпидемиологии и генетики рака, Национальный институт рака, Национальный институт здравоохранения, Bethesda, 20850, штат Мэриленд, США;
(3) Monjoy Saha, Отделение эпидемиологии и генетики рака, Национальный институт рака, Национальный институт здравоохранения, Bethesda, 20850, штат Мэриленд, США;
(4) Джонас С. Алмейда, Отделение эпидемиологии и генетики рака, Национальный институт рака, Национальный институт здравоохранения, Бетесда, 20850, штат Мэриленд, США;
(5) Арлиндо Л. Оливейра, Институт Верхний Течнико, Университет де Лисбоа, ав. Rovisco Pais, Лиссабон, 1049-001, Португалия и INESC-ID, R. Alves Redol 9, Lisbon, 1000-029, Португалия.
Эта статья есть
Оригинал

