Как исследователи предварительно обрабатывают гигабайт WSIS для глубокого обучения
17 июля 2025 г.Таблица ссылок
Аннотация и I. Введение
Материалы и методы
2.1. Несколько экземпляров обучения
2.2. Модель архитектуры
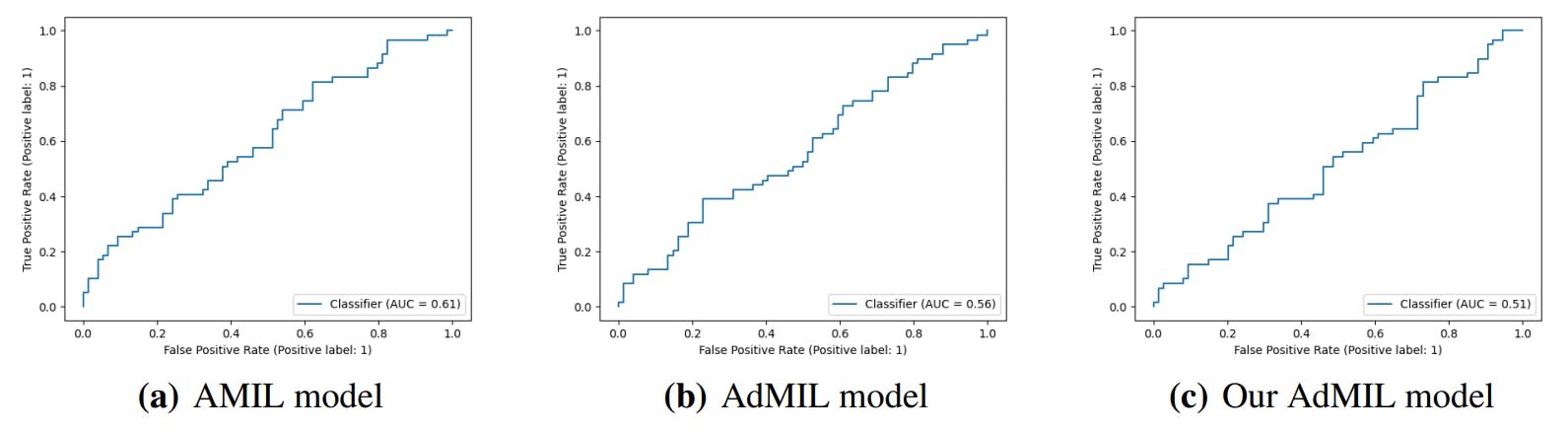
Результаты
3.1. Методы обучения
3.2. Наборы данных
3.3. WSI предварительно обработайте трубопровод
3.4. Результаты классификации и обнаружения ROI
Дискуссия
4.1. Задача обнаружения опухоли
4.2. Задача обнаружения мутаций генов
Выводы
Благодарности
Авторская декларация и ссылки
3.3. WSI предварительно обработайте трубопровод
WSI часто слишком велики, чтобы быть непосредственно обрабатываемыми моделями глубокого обучения. Слайды могут занимать более 6 ГБ памяти, поэтому в нашем случае наличие набора набора необработанных слайдов невозможно. Чтобы сохранить наш набор данных на местном уровне, мы решили сосредоточиться на одном увеличении одновременно. Все наши модели были обучены отдельным уровням увеличения, что гарантировало, что это не будет проблемой. Для некоторых увеличений общее количество патчей может превышать доступное место для хранения. Чтобы преодолеть эту проблему, мы разработали трубопровод, который получает плитки WSI, процессы, фильтры, эффективно кодирует их, и сохраняет полученные встроенные вкладыши и соответствующие метаданные в файлах HDF5. Этот трубопровод объясняется в следующих разделах.
3.3.1. Извлечение метаданных WSI
Мы начнем с извлечения информации о каждом слайде: ID, тип слайда меток и микрон на пиксель (MPP), при котором слайд был изначально сканирован. В конце концов, все извлеченные метаданные сохраняются в файле CSV (разделенные запятыми значениями) для дальнейшей обработки.
3.3.2. Плата и предварительная обработка
Целые слайд -изображения могут иметь много патчей, состоящих исключительно из фона или артефактов, которые делают их непригодными для обучения наших моделей. Извлечение всех этих ненужных пятен и проверка их один за другим становится неэффективным и трудоемким. Чтобы избежать этого, мы используем многомасштабное свойство WSI, чтобы избежать извлечения пятен, которые определенно будут содержать только фон.
Плитка на уровне миниатюры первоначально извлекается. К нему применяется пороговое значение [16] [16], а также тесная морфологическая операция для фильтрации шума. Затем мы извлекаем полученные координаты черного пикселя, получая набор пикселей 𝑃, пиксели, которые соответствуют ткани.
При приобретении WSI часто существуют непреднамеренные артефакты из -за ручного подготовки тканей, окрашивания и сканирования оборудования, а также аннотаций патологов. Чтобы смягчить количество плиток, содержащих артефакты, а также удалить некоторые фоновые пиксели, которые могли проходить через предыдущий фильтр, цвет каждого пикселя 𝑝 ∈ 𝑃 сравнивал со средним цветом 𝑃, путем расчета их евклидового расстояния и сравнения его с предварительно определенным порогом. Координаты пикселей, которые выполняли это условие, были затем сохранялись и использовались для расчета соответствующих координат пятен при желаемом увеличении, используя иерархические свойства WSI и метаданные, извлеченные с шага в 3.3.1.
Поскольку эти фильтры были применены на уровне миниатюры, некоторые плитки все еще не подходили для окончательного набора данных. После получения каждой плитки мы проверили, был ли ее размер в координатах изображения составлял 512 × 512 px. Если он был меньше, он был соответствующим образом сочетается со средним цветом фона. Процент ткани, присутствующей в плите, затем рассчитывали и сравнивали с порогом. Если он не содержит достаточно ткани, изображение отбрасывается.
Для обнаружения мутаций генов, из -за размера слайдов FFPE и используемых уровней увеличения, мы применили случайную выборку на плитки:
• При увеличении в 5 раза мы выбрали 60% отфильтрованных плиток, когда их число было больше, чем определенный предел; В противном случае мы использовали все плитки.
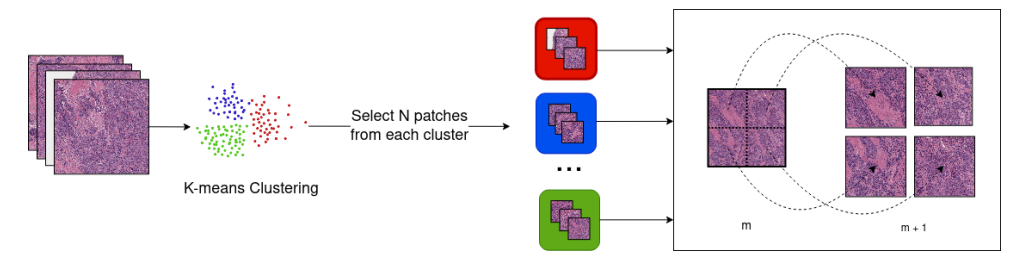
• В случае увеличения 10x и 20x мы применили кластеризацию на плитки от предыдущего увеличения и отобрали выбранный номер N плиток на кластер. Затем мы приступили к использованию иерархических свойств WSI для извлечения соответствующих плиток в желаемых увеличениях. Например, мы выполнили кластеризацию K-средних на плитках при увеличении 5x, отобрали максимум 20 плиток из каждого кластера и продолжали извлекать для каждой плитки соответствующие плитки при увеличении 10-кратного увеличения (рис. 2).
3.3.3. Извлечение функций
Как упоминалось ранее, из плитки были созданы встроения. Для этого мы выбрали Kimianet [18], Densenet 121, предварительно подготовленный для классификации подтипа опухоли WSI, которая производит встраивающие векторы с длиной 1024. Эта модель была обучена исключительно на слайдах FFPE.
Мы также выполнили два увеличения данных на плитку, состоящие из случайного возмущения пятен хед, добавления шума гаусса, вращений и горизонтальных и вертикальных переворот. Встроения были сгенерированы и для этих дополненных плиток. Затем встраивания сохраняются в файле HDF5, а также соответствующие соответствующие метаданные, такие как координаты патчей и метки. Этот файл затем можно быстро прочитать в памяти во время учебного процесса моделей.
Из-за размера набора данных немедленно преобразование каждого пятна 512 × 512 пикселей в внедрение длины 1024 сохраняет пространство для хранения (с уменьшением размера, близкого к тысячу раз) и времени для обучения модели и позволяет нам иметь целый набор данных локально, вместо того, чтобы приносить данные каждый раз, нам необходимы для обучения или тонкого набора моделей. Кроме того, немедленно представляя слайды в пространстве функций, в отличие от пиксельного пространства, мы смогли одновременно вписать все патчи в слайде в память графического процессора, что особенно полезно для множества подходов к обучению.
Авторы:
(1) Мармим Афонсо, институт Superior Técnico, Universidade de Lisboa, Av. Ровиско Паис, Лиссабон, 1049-001, Португалия;
(2) Praphulla M.S. Bhawsar, Отдел эпидемиологии и генетики рака, Национальный институт рака, Национальный институт здравоохранения, Bethesda, 20850, штат Мэриленд, США;
(3) Monjoy Saha, Отделение эпидемиологии и генетики рака, Национальный институт рака, Национальный институт здравоохранения, Bethesda, 20850, штат Мэриленд, США;
(4) Джонас С. Алмейда, Отделение эпидемиологии и генетики рака, Национальный институт рака, Национальный институт здравоохранения, Бетесда, 20850, штат Мэриленд, США;
(5) Арлиндо Л. Оливейра, Институт Верхний Течнико, Университет де Лисбоа, ав. Rovisco Pais, Лиссабон, 1049-001, Португалия и INESC-ID, R. Alves Redol 9, Lisbon, 1000-029, Португалия.
Эта статья есть
Оригинал
